问题导读
1.本文执行聚类包括哪三个MR任务?
2.画决策图的作用是什么?
3.执行分类的思路是什么?

接上篇:
Hadoop Web项目--Friend Find系统(1)
6. 最佳DC
最佳DC是在”聚类算法“-->”执行聚类“时使用的参数,具体可以参考Clustering by fast search and find of density peaks相关论文。
在寻找最佳DC时是把所有距离按照从大到小进行排序,然后顺序遍历这些距离,取前面的2%左右的数据。这里排序由于在”计算距离“MR任务时,已经利用其Map->reduce的排序性即可,其距离已经按照距离的大小从小到大排序了,所以只需遍历即可,这里使用直接遍历序列文件的方式,如下:
[mw_shl_code=java,true]/**
* 根据给定的阈值百分比返回阈值
*
* @param percent
* 一般为1~2%
* @return
*/
public static double findInitDC(double percent, String path,long iNPUT_RECORDS2) {
Path input = null;
if (path == null) {
input = new Path(HUtils.getHDFSPath(HUtils.FILTER_CALDISTANCE
+ "/part-r-00000"));
} else {
input = new Path(HUtils.getHDFSPath(path + "/part-r-00000"));
}
Configuration conf = HUtils.getConf();
SequenceFile.Reader reader = null;
long counter = 0;
long percent_ = (long) (percent * iNPUT_RECORDS2);
try {
reader = new SequenceFile.Reader(conf, Reader.file(input),
Reader.bufferSize(4096), Reader.start(0));
DoubleWritable dkey = (DoubleWritable) ReflectionUtils.newInstance(
reader.getKeyClass(), conf);
Writable dvalue = (Writable) ReflectionUtils.newInstance(
reader.getValueClass(), conf);
while (reader.next(dkey, dvalue)) {// 循环读取文件
counter++;
if(counter%1000==0){
Utils.simpleLog("读取了"+counter+"条记录。。。");
}
if (counter >= percent_) {
HUtils.DELTA_DC = dkey.get();// 赋予最佳DC阈值
break;
}
}
} catch (Exception e) {
e.printStackTrace();
} finally {
IOUtils.closeStream(reader);
}
return HUtils.DELTA_DC;
}
[/mw_shl_code]
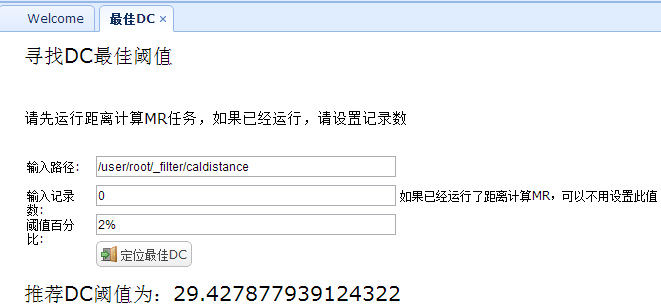
这里需要说明一下,经过试验,发现使用距离阈值29.4时,聚类的决策图中的聚类中心向量并不是十分明显,所以在下面使用的阈值是100;
4.3 聚类算法1. 执行聚类
执行聚类包括三个MR任务:局部密度MR、最小距离MR以及排序MR任务:
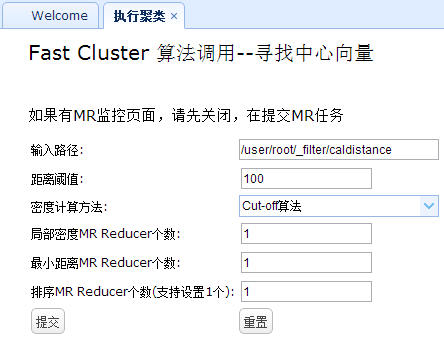
1)局部密度MR
局部密度计算使用的输入文件即是前面计算的距离文件,其MR数据流如下:
[mw_shl_code=java,true]/**
* Find the local density of every point vector
*
* 输入为 <key,value>--> <distance,<id_i,id_j>>
* <距离,<向量i编号,向量j编号>>
*
* Mapper:
* 输出向量i编号,1
* 向量j编号,1
* Reducer:
* 输出
* 向量i编号,局部密度
* 有些向量是没有局部密度的,当某个向量距离其他点的距离全部都大于给定阈值dc时就会发生
* @author fansy
* @date 2015-7-3
*/[/mw_shl_code]
Mapper的逻辑如下:
[mw_shl_code=java,true]/**
* 输入为<距离d_ij,<向量i编号,向量j编号>>
* 根据距离dc阈值判断距离d_ij是否小于dc,符合要求则
* 输出
* 向量i编号,1
* 向量j编号,1
* @author fansy
* @date 2015-7-3
*/[/mw_shl_code]
map函数:
[mw_shl_code=java,true]public void map(DoubleWritable key,IntPairWritable value,Context cxt)throws InterruptedException,IOException{
double distance= key.get();
if(method.equals("gaussian")){
one.set(Math.pow(Math.E, -(distance/dc)*(distance/dc)));
}
if(distance<dc){
vectorId.set(value.getFirst());
cxt.write(vectorId, one);
vectorId.set(value.getSecond());
cxt.write(vectorId, one);
}
}[/mw_shl_code]
这里的密度有两种计算方式,根据前台传入的参数选择不同的算法即可,这里默认使用的cut-off,即局部密度有一个点则局部密度加1;
reducer中的reduce逻辑即把相同的点的局部密度全部加起来即可:
[mw_shl_code=java,true]public void reduce(IntWritable key, Iterable<DoubleWritable> values,Context cxt)
throws IOException,InterruptedException{
double sum =0;
for(DoubleWritable v:values){
sum+=v.get();
}
sumAll.set(sum);//
cxt.write(key, sumAll);
Utils.simpleLog("vectorI:"+key.get()+",density:"+sumAll);
}[/mw_shl_code]
2)最小距离MR
最小距离MR逻辑如下:
[mw_shl_code=java,true]/**
* find delta distance of every point
* 寻找大于自身密度的最小其他向量的距离
* mapper输入:
* 输入为<距离d_ij,<向量i编号,向量j编号>>
* 把LocalDensityJob的输出
* i,density_i
* 放入一个map中,用于在mapper中进行判断两个局部密度的大小以决定是否输出
* mapper输出:
* i,<density_i,min_distance_j>
* IntWritable,DoublePairWritable
* reducer 输出:
* <density_i*min_distancd_j> <density_i,min_distance_j,i>
* DoubleWritable, IntDoublePairWritable
* @author fansy
* @date 2015-7-3
*/[/mw_shl_code]
这里reducer输出为每个点(即每个用户)局部密度和最小距离的乘积,一种方式寻找聚类中心个数的方法就是把这个乘积从大到小排序,并把这些点画折线图,看其斜率变化最大的点,取前面点的个数即为聚类中心个数。
3)排序MR
排序MR即把2)的局部密度和最小距离的乘积进行排序,这里可以利用map-reduce的排序性,自定义一个Writable,然后让其按照值的大小从大到小排序。
[mw_shl_code=java,true]/**
*
*/
package com.fz.fastcluster.keytype;
/**
* 自定义DoubleWritable
* 修改其排序方式,
* 从大到小排列
* @author fansy
* @date 2015-7-3
*/
import java.io.DataInput;
import java.io.DataOutput;
import java.io.IOException;
import org.apache.hadoop.classification.InterfaceAudience;
import org.apache.hadoop.classification.InterfaceStability;
import org.apache.hadoop.io.WritableComparable;
import org.apache.hadoop.io.WritableComparator;
/**
* Writable for Double values.
*/
@InterfaceAudience.Public
@InterfaceStability.Stable
public class CustomDoubleWritable implements WritableComparable<CustomDoubleWritable> {
private double value = 0.0;
public CustomDoubleWritable() {
}
public CustomDoubleWritable(double value) {
set(value);
}
@Override
public void readFields(DataInput in) throws IOException {
value = in.readDouble();
}
@Override
public void write(DataOutput out) throws IOException {
out.writeDouble(value);
}
public void set(double value) { this.value = value; }
public double get() { return value; }
/**
* Returns true iff <code>o</code> is a DoubleWritable with the same value.
*/
@Override
public boolean equals(Object o) {
if (!(o instanceof CustomDoubleWritable)) {
return false;
}
CustomDoubleWritable other = (CustomDoubleWritable)o;
return this.value == other.value;
}
@Override
public int hashCode() {
return (int)Double.doubleToLongBits(value);
}
@Override
public int compareTo(CustomDoubleWritable o) {// 修改这里即可
return (value < o.value ? 1 : (value == o.value ? 0 : -1));
}
@Override
public String toString() {
return Double.toString(value);
}
/** A Comparator optimized for DoubleWritable. */
public static class Comparator extends WritableComparator {
public Comparator() {
super(CustomDoubleWritable.class);
}
@Override
public int compare(byte[] b1, int s1, int l1,
byte[] b2, int s2, int l2) {
double thisValue = readDouble(b1, s1);
double thatValue = readDouble(b2, s2);
return (thisValue < thatValue ? 1 : (thisValue == thatValue ? 0 : -1));
}
}
static { // register this comparator
WritableComparator.define(CustomDoubleWritable.class, new Comparator());
}
}
[/mw_shl_code]
2. 画决策图
画决策图,直接解析云平台的排序MR的输出,然后取前面的500条记录(前面500条记录包含的局部密度和最小距离的乘积的最大的500个,后面的点更不可能成为聚类中心点,所以这里只取500个,同时需要注意,如果前面设置排序MR的reducer个数大于一个,那么其输出为多个文件,则这里是取每个文件的前面500个向量)
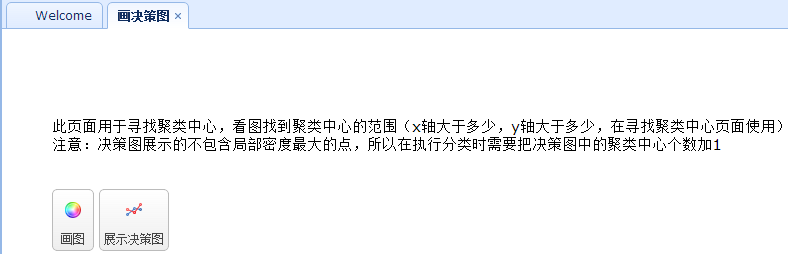

依次点击画图,展示决策图,即可看到画出的决策图:
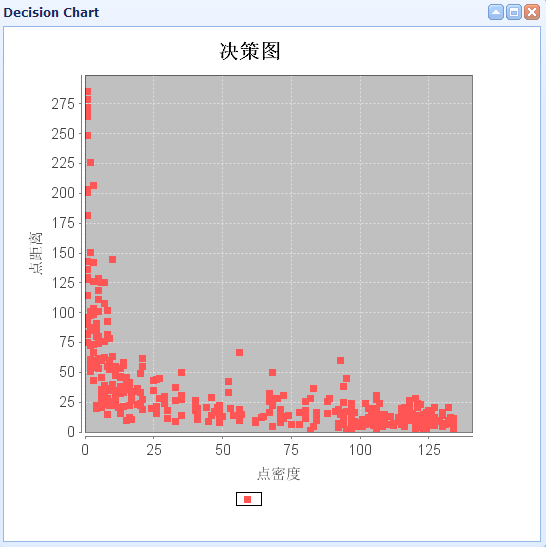
聚类中心应该是取右上角位置的点,所以这里选择去点密度大于50,点距离大于50的点,这里有3个,加上没有画出来的局部密度最大的点,一共有4个聚类中心向量。
3. 寻找聚类中心
寻找聚类中心就是根据前面决策图得到的点密度和点距离阈值来过滤排序MR的输出,得到符合要求的用户ID,这些用户ID即是聚类中心向量的ID。接着,根据这些ID在数据库中找到每个用户ID对应的有效向量(reputation,upVotes,downVotes,views)写入HDFS和本地文件。写入HDFS是为了作为分类的中心点,写入本地是为了后面查看的方便。
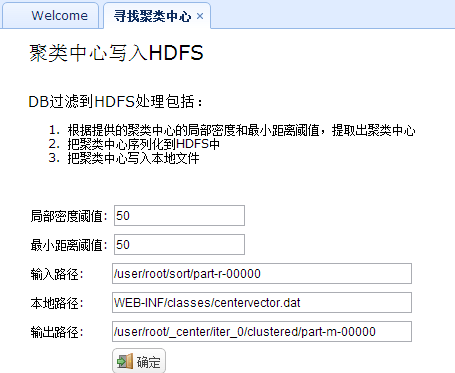
代码如下:
[mw_shl_code=java,true]/**
* 根据给定的阈值寻找聚类中心向量,并写入hdfs
* 非MR任务,不需要监控,注意返回值
*/
public void center2hdfs(){
// localfile:method
// 1. 读取SortJob的输出,获取前面k条记录中的大于局部密度和最小距离阈值的id;
// 2. 根据id,找到每个id对应的记录;
// 3. 把记录转为double[] ;
// 4. 把向量写入hdfs
// 5. 把向量写入本地文件中,方便后面的查看
Map<String,Object> retMap=new HashMap<String,Object>();
Map<Object,Object> firstK =null;
List<Integer> ids= null;
List<UserData> users=null;
try{
firstK=HUtils.readSeq(input==null?HUtils.SORTOUTPUT+"/part-r-00000":input,
100);// 这里默认使用 前100条记录
ids=HUtils.getCentIds(firstK,numReducerDensity,numReducerDistance);
// 2
users = dBService.getTableData("UserData",ids);
Utils.simpleLog("聚类中心向量有"+users.size()+"个!");
// 3,4,5
HUtils.writecenter2hdfs(users,method,output);
}catch(Exception e){
e.printStackTrace();
retMap.put("flag", "false");
retMap.put("msg", e.getMessage());
Utils.write2PrintWriter(JSON.toJSONString(retMap));
return ;
}
retMap.put("flag", "true");
Utils.write2PrintWriter(JSON.toJSONString(retMap));
return ;
}[/mw_shl_code]
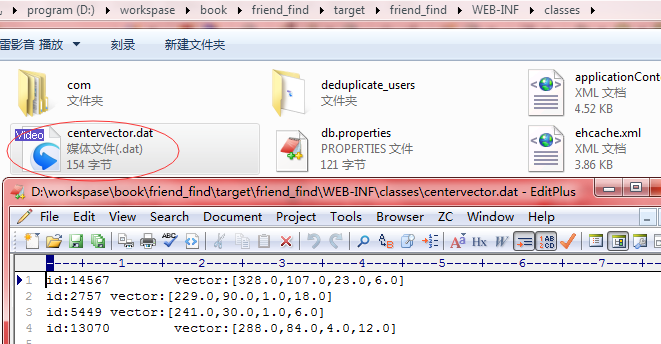
写入HDFS和本地的聚类中心如下:
4. 执行分类
4.1 执行分类的思路为:
1)聚类中心向量已经写入到_center/iter_0/clustered/part-m-00000中;接着,拷贝原始用户向量(即“DB过滤到HDFS”的输出)到_center/iter_0/unclustered/
2)执行第一次分类,使用Mapper即可,mapper逻辑为读取_center/iter_0/unclustered/里面的所有文件的每一行,针对每一行A,读取_center/iter_0/clustered/里面所有的数据,循环判断这些向量和A的距离,找到和A的距离最小的距离(同时这个距离需要满足大于给定的阈值),并记录这个距离对应向量的类型type,那么就可以输出向量A和类型type,那向量A就已经被分类了,分类后的数据写入到_center/iter_1/clustered里面;如果没有找到最小距离(即所有的距离都大于给定的阈值),那么向量A就是没有被分类的,那么把数据写入到_center/iter_1/unclustered里面;
3)在2)中的mapper中需要记录分类数据和未分类数据的记录数,这样在MR任务运行完成后,即可根据这两个数值来判断是否需要进行下次循环,如果这两个数值都是零,那么就退出循环,否则进行下一步;
4)在第i次循环(i>=2)时,使用_center/iter_(i-1)/unclustered里面的数据作为输入,针对这个输入的每一行向量A,遍历_center/iter_1/clustered ~ _center/iter_(i-1)/clustered,使用2)中的方式对A进行分类,如果完成分类,那么就把数据写入到_center/iter_i/clustered,否则写入到_center/iter_i/unclustered里面;
5)根据第i次MR任务记录的Clustered和Unclustered的值来判断是否进行下次循环,不用则退出循环,否则继续循环进入4);
map函数代码:
[mw_shl_code=java,true]public void map(IntWritable key,DoubleArrIntWritable value,Context cxt){
double[] inputI= value.getDoubleArr();
// hdfs
Configuration conf = cxt.getConfiguration();
FileSystem fs = null;
Path path = null;
SequenceFile.Reader reader = null;
try {
fs = FileSystem.get(conf);
// read all before center files
String parentFolder =null;
double smallDistance = Double.MAX_VALUE;
int smallDistanceType=-1;
double distance;
// if iter_i !=0,then start i with 1,else start with 0
for(int i=start;i<iter_i;i++){// all files are clustered points
parentFolder=HUtils.CENTERPATH+"/iter_"+i+"/clustered";
RemoteIterator<LocatedFileStatus> files=fs.listFiles(new Path(parentFolder), false);
while(files.hasNext()){
path = files.next().getPath();
if(!path.toString().contains("part")){
continue; // return
}
reader = new SequenceFile.Reader(conf, Reader.file(path),
Reader.bufferSize(4096), Reader.start(0));
IntWritable dkey = (IntWritable) ReflectionUtils.newInstance(
reader.getKeyClass(), conf);
DoubleArrIntWritable dvalue = (DoubleArrIntWritable) ReflectionUtils.newInstance(
reader.getValueClass(), conf);
while (reader.next(dkey, dvalue)) {// read file literally
distance = HUtils.getDistance(inputI, dvalue.getDoubleArr());
if(distance>dc){// not count the farest point
continue;
}
// 这里只要找到离的最近的点并且其distance<=dc 即可,把这个点的type赋值给当前值即可
if(distance<smallDistance){
smallDistance=distance;
smallDistanceType=dvalue.getIdentifier();
}
}// while
}// while
}// for
vectorI.set(key.get());// 用户id
typeDoubleArr.setValue(inputI,smallDistanceType);
if(smallDistanceType!=-1){
log.info("clustered-->vectorI:{},typeDoubleArr:{}",new Object[]{vectorI,typeDoubleArr.toString()});
cxt.getCounter(ClusterCounter.CLUSTERED).increment(1);
out.write("clustered", vectorI, typeDoubleArr,"clustered/part");
}else{
log.info("unclustered---->vectorI:{},typeDoubleArr:{}",new Object[]{vectorI,typeDoubleArr.toString()});
cxt.getCounter(ClusterCounter.UNCLUSTERED).increment(1);
out.write("unclustered", vectorI, typeDoubleArr,"unclustered/part");
}
} catch (Exception e) {
e.printStackTrace();
} finally {
IOUtils.closeStream(reader);
}
}[/mw_shl_code]
4.2 执行分类的阈值设置
每次循环执行分类时,阈值都是变化的,这里采取的方式是:
1. 计算聚类中心向量两两之间的距离,并按照距离排序,从小到大,每次循环取出距离的一半当做阈值,一直取到最后一个距离;
2. 当进行到K*(K-1)/2个距离时,即最后一个距离(K个聚类中心向量)后,下次循环的阈值设置为当前阈值翻倍,即乘以2;并计数,当再循环k次后,此阈值将不再变化;
3. 这样设置可以减少误判,同时控制循环的次数;
[mw_shl_code=java,true]public void run() {
input=input==null?HUtils.FILTER_PREPAREVECTORS:input;
// 删除iter_i(i>0)的所有文件
try {
HUtils.clearCenter((output==null?HUtils.CENTERPATH:output));
} catch (FileNotFoundException e2) {
e2.printStackTrace();
} catch (IOException e2) {
e2.printStackTrace();
}
output=output==null?HUtils.CENTERPATHPREFIX:output+"/iter_";
// 加一个操作,把/user/root/preparevectors里面的数据复制到/user/root/_center/iter_0/unclustered里面
HUtils.copy(input,output+"0/unclustered");
try {
Thread.sleep(200);// 暂停200ms
} catch (InterruptedException e1) {
e1.printStackTrace();
}
// 求解dc的阈值,这里的dc不用传入进来即可,即delta的值
// 阈值问题可以在讨论,这里暂时使用传进来的阈值即可
// double dc =dcs[0];
// 读取聚类中心文件
Map<Object,Object> vectorsMap= HUtils.readSeq(output+"0/clustered/part-m-00000", Integer.parseInt(k));
double[][] vectors = HUtils.getCenterVector(vectorsMap);
double[] distances= Utils.getDistances(vectors);
// 这里不使用传入进来的阈值
int iter_i=0;
int ret=0;
double tmpDelta=0;
int kInt = Integer.parseInt(k);
try {
do{
if(iter_i>=distances.length){
// delta= String.valueOf(distances[distances.length-1]/2);
// 这里使用什么方式还没有想好。。。
// 使用下面的方式
tmpDelta=Double.parseDouble(delta);
while(kInt-->0){// 超过k次后就不再增大
tmpDelta*=2;// 每次翻倍
}
delta=String.valueOf(tmpDelta);
}else{
delta=String.valueOf(distances[iter_i]/2);
}
log.info("this is the {} iteration,with dc:{}",new Object[]{iter_i,delta});
String[] ar={
HUtils.getHDFSPath(output)+iter_i+"/unclustered",
HUtils.getHDFSPath(output)+(iter_i+1),//output
//HUtils.getHDFSPath(HUtils.CENTERPATHPREFIX)+iter_i+"/clustered/part-m-00000",//center file
k,
delta,
String.valueOf((iter_i+1))
};
try{
ret = ToolRunner.run(HUtils.getConf(), new ClusterDataJob(), ar);
if(ret!=0){
log.info("ClusterDataJob failed, with iteration {}",new Object[]{iter_i});
break;
}
}catch(Exception e){
e.printStackTrace();
}
iter_i++;
HUtils.JOBNUM++;// 每次循环后加1
}while(shouldRunNextIter());
} catch (IllegalArgumentException e) {
e.printStackTrace();
}
if(ret==0){
log.info("All cluster Job finished with iteration {}",new Object[]{iter_i});
}
}[/mw_shl_code]
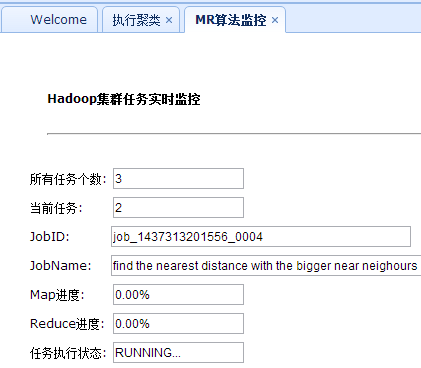
4.3 执行分类的监控思路
执行监控还是使用之前的代码,但是这里的MR任务个数一开始并不能直接确定,那就不能控制监控循环结束的时间。所以这里需要进行修改,这里在MR任务循环完成之后,设置JOBNUM的值来控制监控任务的结束,并且一开始设置JOBNUM为2,这样在一开始的MR运行结束后就会进行下一次监控循环(这里有个假设就是监控不会只有一次),并且在MR任务每次结束后JOBNUM的值需要递增1:
[mw_shl_code=java,true]public void runCluster2(){
Map<String ,Object> map = new HashMap<String,Object>();
try {
//提交一个Hadoop MR任务的基本流程
// 1. 设置提交时间阈值,并设置这组job的个数
//使用当前时间即可,当前时间往前10s,以防服务器和云平台时间相差
HUtils.setJobStartTime(System.currentTimeMillis()-10000);//
// 由于不知道循环多少次完成,所以这里设置为2,每次循环都递增1
// 当所有循环完成的时候,就该值减去2即可停止监控部分的循环
HUtils.JOBNUM=2;
// 2. 使用Thread的方式启动一组MR任务
new Thread(new RunCluster2(input, output,delta, record)).start();
// 3. 启动成功后,直接返回到监控,同时监控定时向后台获取数据,并在前台展示;
map.put("flag", "true");
map.put("monitor", "true");
} catch (Exception e) {
e.printStackTrace();
map.put("flag", "false");
map.put("monitor", "false");
map.put("msg", e.getMessage());
}
Utils.write2PrintWriter(JSON.toJSONString(map));
}[/mw_shl_code]
在MR任务循环结束后,重新设置JOBNUM的值即可控制监控的循环停止:
[mw_shl_code=java,true]/**
* 是否应该继续下次循环
* 直接使用分类记录数和未分类记录数来判断
* @throws IOException
* @throws IllegalArgumentException
*/
private boolean shouldRunNextIter() {
if(HUtils.UNCLUSTERED==0||HUtils.CLUSTERED==0){
HUtils.JOBNUM-=2;// 不用监控 则减去2;
return false;
}
return true;
}[/mw_shl_code]
执行分类页面:
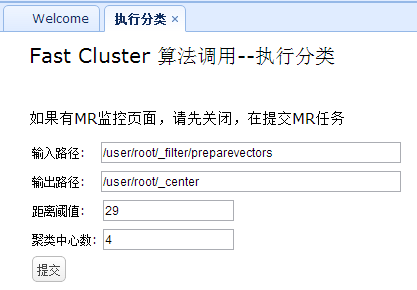
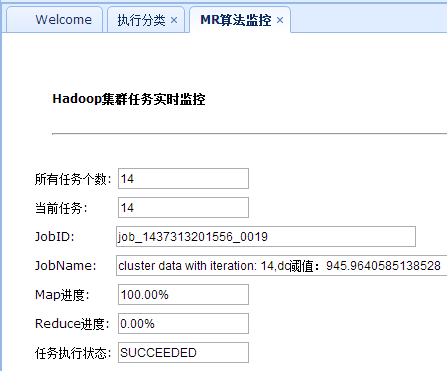
这里距离阈值是没有用的,后台直接使用上面的算法得到;循环完成监控界面的最终阈值,如下所示:
4.4 聚类中心及推荐1. 组别入库
组别入库,即是把_center/iter_i/clustered里面的数据解析导入数据库中,导入数据库还是使用上面的批插入操作:
[mw_shl_code=java,true]/**
* 把分类的数据解析到list里面
* @param path
* @return
*/
private static Collection<? extends UserGroup> resolve(Path path) {
// TODO Auto-generated method stub
List<UserGroup> list = new ArrayList<UserGroup>();
Configuration conf = HUtils.getConf();
SequenceFile.Reader reader = null;
int i=0;
try {
reader = new SequenceFile.Reader(conf, Reader.file(path),
Reader.bufferSize(4096), Reader.start(0));
IntWritable dkey = (IntWritable) ReflectionUtils
.newInstance(reader.getKeyClass(), conf);
DoubleArrIntWritable dvalue = (DoubleArrIntWritable) ReflectionUtils
.newInstance(reader.getValueClass(), conf);
while (reader.next(dkey, dvalue)) {// 循环读取文件
// 使用这个进行克隆
list.add(new UserGroup(i++,dkey.get(),dvalue.getIdentifier()));
}
} catch (Exception e) {
e.printStackTrace();
} finally {
IOUtils.closeStream(reader);
}
Utils.simpleLog("读取"+list.size()+"条记录,文件:"+path.toString());
return list;
}[/mw_shl_code]
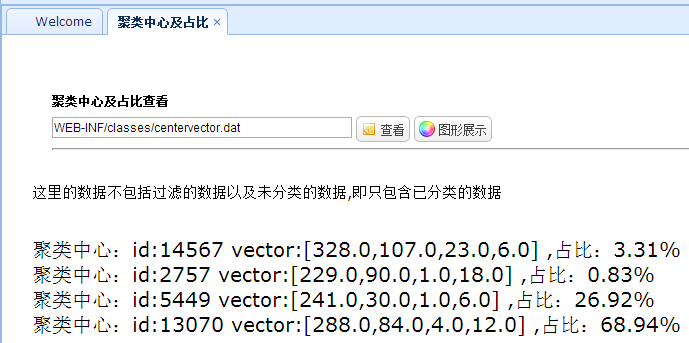
2. 聚类中心及占比
聚类中心及占比直接使用数据库中的数据进行统计的,即统计1.中的分类数据每个类别的总记录数,然后再进行计算。聚类中心即直接读取之前写入本地的聚类中心向量文件即可。
3. 用户查询及推荐
用户查询及推荐即使用用户组内的用户来进行推荐。根据给定的ID来查询该用户的分组,如果有分组,那么就查询出该分组内的用户,展示到前台:
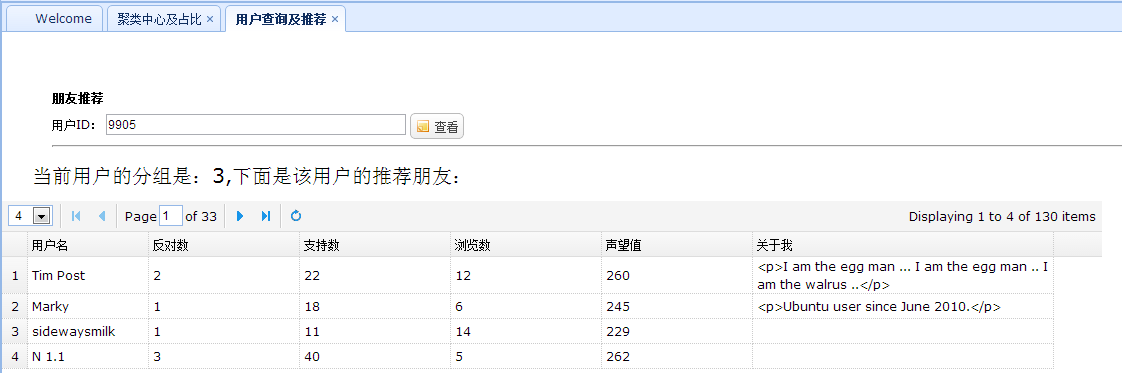
5. 总结1. 原始数据经过去重、过滤后,仅剩下541条记录,即对541条记录进行聚类,不算大数据处理;
2. 上面的Hadoop实现的聚类算法,可以使用大数据来测试下,看下效果;
3. 聚类算法在计算两两之间的距离时随着文件的增大,其耗时增长很快O(N^2);
4. 使用组内的用户来对用户直接进行推荐的方式有待商榷,可以考虑在组内使用其他方式来过滤用户(比如根据地理位置等信息,该信息在原始数据中是有的),再次推荐;
5. 本项目仅供学习参考,重心在技术和处理方式以及实现方式上;
分享,成长,快乐 脚踏实地,专注
|
 /2
/2 