问题导读
1.Hadoop实现聚类快速搜索的思路是什么?
2.计算“局部密度距离”中,Mapper实现了什么功能?
3.如何实现根据决策图人工确定聚类个数?

Hadoop版本:2.6.0,Myeclipse:10.0
代码可在https://github.com/fansy1990/fast_cluster下载。
1. 实现思路
1.1. 输入数据
代码可以采用任意维度的数据,使用文本存储,其一般格式如下(为方便作图,使用两位数据):
[mw_shl_code=bash,true]6.5,18.9,2
6.6,17.65,2
6.65,18.3,2
7.3,18.35,2
7.85,18.3,2
7.15,17.8,2
7.6,17.7,2
。。。[/mw_shl_code]
1.2. 整体思路描述
1)计算每个点的“局部密度距离”(参考论文);
2)计算每个点的“超密度最小距离”(这里翻译有点问题,额可以参考原论文);
3)画出决策图来人工判断可以选取的聚类中心个数;
4)根据选择的聚类中心来使用临近点判别法循环使用已分类数据来分类未分类数据;
1.3. 计算“局部密度距离”
计算“局部密度距离”时,遍历输入文件,同时把输入文件当做参考。针对输入文件的每行数据inputI遍历所有输入文件(不包括inputI)来计算其和其他点的距离,如果小于给定的阈值dc,那么就把这个点的局部密度加1(这里计算局部密度有两种方式,参考博客前面给出的参考博客);其Mapper如下:
[mw_shl_code=java,true]package fz.fast_cluster.mr;
import java.io.BufferedReader;
import java.io.IOException;
import java.io.InputStream;
import java.io.InputStreamReader;
import org.apache.hadoop.fs.FileSystem;
import org.apache.hadoop.fs.Path;
import org.apache.hadoop.io.DoubleWritable;
import org.apache.hadoop.io.LongWritable;
import org.apache.hadoop.io.Text;
import org.apache.hadoop.mapreduce.Mapper;
import fz.fast_cluster.keytype.DoubleArrWritable;
import fz.utils.HUtils;
/**
* @author fansy
* @date 2015-5-28
*/
public class LocalDensityMapper extends Mapper<LongWritable, Text, DoubleArrWritable, DoubleWritable> {
private double dc;
private Path input;
private String splitter=",";
private String method =null;
private DoubleWritable sumAll= new DoubleWritable();
private DoubleArrWritable keyDoubleArr;
@Override
public void setup(Context cxt){
dc=cxt.getConfiguration().getDouble("DC", 0);
input=new Path(cxt.getConfiguration().get("INPUT"));//
splitter = cxt.getConfiguration().get("SPLITTER", ",");
method = cxt.getConfiguration().get("METHOD", "gaussian");
}
@Override
public void map(LongWritable key,Text value,Context cxt)throws InterruptedException,IOException{
// get the ith line of all data;
double[] inputI= HUtils.getInputI(value,splitter);
double sum=0;
// hdfs
try{
FileSystem fs = FileSystem.get(cxt.getConfiguration());
InputStream in = fs.open(input);
BufferedReader buff = new BufferedReader(new InputStreamReader(in));
String line = null;
while((line=buff.readLine())!=null){
double[] inputLine = HUtils.getInputI(line,splitter);
double distance = HUtils.getDistance(inputI,inputLine);
if(distance<dc&&distance>0){ // distance should be grater than 0,
if(method.equals("gaussian")){
sum+=Math.pow(Math.E, -(distance/dc)*(distance/dc));
}else{
sum+=1;
}
}
}
}catch(Exception e){
e.printStackTrace();
}
// output if the point has at least one neighbor
if(sum>0){
sumAll.set(sum);
keyDoubleArr = new DoubleArrWritable(inputI);
cxt.write(keyDoubleArr, sumAll);
}
}
}[/mw_shl_code]
1.4. 计算“超密度最小距离”
根据1.3. 计算的结果,使用1.3.的输出作为输入,针对单个输入,遍历1.3.的所有输出并计算点之间的距离,找到距离最小的点,同时要求该距离最小的点对应的点的局部密度要大于当前点的局部密度。针对全局最大的局部密度的点,直接计算距离与其最远的距离即可。其Mapper代码如下:
[mw_shl_code=bash,true]package fz.fast_cluster.mr;
import java.io.IOException;
import org.apache.hadoop.conf.Configuration;
import org.apache.hadoop.fs.Path;
import org.apache.hadoop.io.DoubleWritable;
import org.apache.hadoop.io.IOUtils;
import org.apache.hadoop.io.SequenceFile;
import org.apache.hadoop.io.SequenceFile.Reader;
import org.apache.hadoop.mapreduce.Mapper;
import org.apache.hadoop.util.ReflectionUtils;
import org.slf4j.Logger;
import org.slf4j.LoggerFactory;
import fz.fast_cluster.keytype.DDoubleWritable;
import fz.fast_cluster.keytype.DoubleArrWritable;
import fz.utils.HUtils;
/**
*
* @author fansy
* @date 2015-6-1
*/
public class DeltaDistanceMapper extends
Mapper<DoubleArrWritable, DoubleWritable, DoubleArrWritable, DDoubleWritable> {
private Logger log = LoggerFactory.getLogger(DeltaDistanceMapper.class);
private Path input ;
private DDoubleWritable sd= new DDoubleWritable();
@Override
public void setup(Context cxt){
input = new Path(cxt.getConfiguration().get("INPUT"));
}
// only read for one file
// maybe add more files support for the another time
@Override
public void map(DoubleArrWritable key, DoubleWritable value, Context cxt) throws IOException,InterruptedException{
// hdfs
Configuration conf = cxt.getConfiguration();
SequenceFile.Reader reader = null;
double minDistance = Double.MAX_VALUE;
double maxDistance = -Double.MAX_VALUE;
try {
reader = new SequenceFile.Reader(conf,Reader.file(input),
Reader.bufferSize(4096),Reader.start(0));
DoubleArrWritable dkey = (DoubleArrWritable) ReflectionUtils.newInstance(
reader.getKeyClass(), conf);
DoubleWritable dvalue = (DoubleWritable) ReflectionUtils.newInstance(
reader.getValueClass(), conf);
while (reader.next(dkey, dvalue)) {//
double d =HUtils.getDistance(key.getDoubleArr(),dkey.getDoubleArr());
if(d>maxDistance&& d>0){
maxDistance=d;
}
if(value.get()<dvalue.get()){//
if(d<minDistance&&d>0){ // d should be greater than 0
minDistance=d;
}
}
}
} catch(Exception e){
e.printStackTrace();
}finally {
IOUtils.closeStream(reader);
}
// set the biggest point density
log.info("key:{},value.get:{},minDistance:{},maxDistance:{}",new Object[]{key,value.get(),minDistance,maxDistance});
sd.setDistance(minDistance==Double.MAX_VALUE?maxDistance:minDistance);
sd.setSum(value.get());
// log.info("sd.sum:{},sd.distance:{}", new Object[]{value.get(),minDistance});
cxt.write(key, sd);
}
}[/mw_shl_code]
1.5. 根据决策图人工确定聚类个数
这里首先根据决策图确定聚类个数后,需要找到这k个聚类中心对应的点向量。这里使用的方法是使用“局部密度”*“超密度最小距离”相乘的值来排序,取前k个数据对应的数据向量,然后写入HDFS目录。同时返回各个聚类向量之间的距离范围作为循环分类的阈值,为循环进行临近点分类做准备,其代码如下所示:
[mw_shl_code=java,true]/**
* get the cluster center by the given k
* return the dc for next ClusterDataJob
* @param input
* @param output
* @param k
* @throws IOException
*/
public static double[] getCenterVector(String input ,String output,int k) throws IOException{
double [] r= new double [k];
String[] queue = new String[k];
//initialize the r array
for(int i=0;i<k;i++){
r=-Double.MAX_VALUE;
}
Path path = new Path(input);
Configuration conf = HUtils.getConf();
InputStream in =null;
try {
FileSystem fs = FileSystem.get(URI.create(input), conf);
in = fs.open(path);
BufferedReader read = new BufferedReader(new InputStreamReader(in));
String line=null;
int index=-1;
while((line=read.readLine())!=null){
// [5.5,4.2,1.4,0.2] 5,0.3464101615137755
String[] lines = line.split("\t");
String[] sd= lines[1].split(",");
index =findSmallest(r,Double.parseDouble(sd[0])*Double.parseDouble(sd[1]));
if(index !=-1){
r[index]=Double.parseDouble(sd[0])*Double.parseDouble(sd[1]);
queue[index]=lines[0];
}
}
} catch (IOException e) {
e.printStackTrace();
}finally{
try {
in.close();
} catch (IOException e) {
e.printStackTrace();
}
}
// print
double dc =Double.MAX_VALUE;
double dc_max = -Double.MAX_VALUE;
double distance =0.0;
for(int i=0;i<queue.length;i++){
System.out.print("vector:"+queue);
for(int j=i+1;j<queue.length;j++){
distance = HUtils.getDistance(getInputI(queue.substring(1, queue.length()-1), ","),
getInputI(queue[j].substring(1, queue[j].length()-1), ","));
if(distance<dc){
dc = distance ;
}
if(distance>dc_max){
dc_max=distance;
}
}
System.out.print("\tr:"+r+"\n");
}
// write to hdfs
path = new Path(output);
DoubleArrWritable key = null;
IntWritable value = new IntWritable();
SequenceFile.Writer writer =null;
try{
writer =SequenceFile.createWriter(conf,
Writer.file(path),
Writer.keyClass(DoubleArrWritable.class),
Writer.valueClass(value.getClass())
);
for(int i=0;i<queue.length;i++){
key = new DoubleArrWritable(getInputI(queue.substring(1, queue.length()-1), ","));
value.set(i+1);
writer.append(key, value);
}
}finally{
IOUtils.closeStream(writer);
}
return new double[]{dc/5,dc_max/3};
}[/mw_shl_code]
1.6. 临近点分类
这里使用的方法是根据已经分类的数据来分类未分类的数据:针对所有未分类的数据,遍历所有已分类的数据,寻找其与已分类的数据的距离,维护一个大小为k的距离数组,里面的距离只取最小的k个。一个大小为k的类别数组,如果新的距离小于其中的一个就替换,同时修改类别数据对应的值。遍历完成后,对距离数组排序,注意类别数组也要使用距离数据的排序规则(即距离数组和类别数组是一一对一个的)。遍历距离数组,直到第一个类别数组有值type,则返回该数据点以及分类的类别type,如果没有值,则把该数据标记为未分类(即分类不成功,需要下次循环再次分类)。其mapper代码如下:
[mw_shl_code=java,true]package fz.fast_cluster.mr;
import java.io.IOException;
import org.apache.hadoop.conf.Configuration;
import org.apache.hadoop.fs.FileSystem;
import org.apache.hadoop.fs.LocatedFileStatus;
import org.apache.hadoop.fs.Path;
import org.apache.hadoop.fs.RemoteIterator;
import org.apache.hadoop.io.IOUtils;
import org.apache.hadoop.io.IntWritable;
import org.apache.hadoop.io.SequenceFile;
import org.apache.hadoop.io.SequenceFile.Reader;
import org.apache.hadoop.mapreduce.Mapper;
import org.apache.hadoop.mapreduce.lib.output.MultipleOutputs;
import org.apache.hadoop.util.ReflectionUtils;
import org.slf4j.Logger;
import org.slf4j.LoggerFactory;
import fz.fast_cluster.keytype.DoubleArrWritable;
import fz.utils.HUtils;
/**
* @author fansy
* @date 2015-6-2
*/
public class ClusterDataMapper extends Mapper<DoubleArrWritable, IntWritable, DoubleArrWritable, IntWritable> {
private Logger log = LoggerFactory.getLogger(ClusterDataMapper.class);
// private String center = null;
private int k =-1;
private double dc =0.0;
private int iter_i =0;
private int start =0;
private DoubleArrWritable doubleArr;
private IntWritable typeInt = new IntWritable();
private MultipleOutputs<DoubleArrWritable,IntWritable> out;
@Override
public void setup(Context cxt){
// center = cxt.getConfiguration().get("CENTER");
k = cxt.getConfiguration().getInt("K", 3);
dc = cxt.getConfiguration().getDouble("DC", Double.MAX_VALUE);
iter_i=cxt.getConfiguration().getInt("ITER_I", 0);
start=iter_i!=1?1:0;
out = new MultipleOutputs<DoubleArrWritable,IntWritable>(cxt);
}
@Override
public void map(DoubleArrWritable key,IntWritable value,Context cxt){
double[] inputI= key.getDoubleArr();
int[] types = new int[k];
double[] smallDistance = new double[k];// k clustered points near the given inpuI
// initial smallDistance and types
for(int i=0;i<k;i++){
smallDistance=Double.MAX_VALUE;
types=-1;
}
// hdfs
Configuration conf = HUtils.getConf();
FileSystem fs = null;
Path path = null;
SequenceFile.Reader reader = null;
try {
fs = FileSystem.get(conf);
// read all before center files
String parentFolder =null;
double distance = Double.MAX_VALUE;
// if iter_i !=0,then start i with 1,else start with 0
for(int i=start;i<iter_i;i++){// all files are clustered points
parentFolder=HUtils.getHDFSPath(HUtils.CENTERPATH+"/iter_"+i+"/clustered");
RemoteIterator<LocatedFileStatus> files=fs.listFiles(new Path(parentFolder), false);
while(files.hasNext()){
path = files.next().getPath();
if(!path.toString().contains("part")){
continue; // return
}
reader = new SequenceFile.Reader(conf, Reader.file(path),
Reader.bufferSize(4096), Reader.start(0));
DoubleArrWritable dkey = (DoubleArrWritable) ReflectionUtils.newInstance(
reader.getKeyClass(), conf);
IntWritable dvalue = (IntWritable) ReflectionUtils.newInstance(
reader.getValueClass(), conf);
while (reader.next(dkey, dvalue)) {// read file literally
distance = HUtils.getDistance(inputI, dkey.getDoubleArr());
if(distance>=dc){// not count the farest point
continue;
}
// else if distance is small enough than modify the small distance and the type
checkAndModify(smallDistance,types,distance ,dvalue.get());
}
}
}
// else
log.info("smallDistance:{},types:{}",new Object[]{HUtils.doubleArr2Str(smallDistance),
HUtils.intArr2Str(types)});
int typeIndex = getTypeIndex(smallDistance,types);
log.info("smallDistance:{},types:{}",new Object[]{HUtils.doubleArr2Str(smallDistance),
HUtils.intArr2Str(types)});
doubleArr = new DoubleArrWritable(inputI);
typeInt.set(typeIndex);
if(typeIndex!=-1){
log.info("clustered-->doubleArr:{},typeInt:{}",new Object[]{doubleArr,typeInt});
out.write("clustered", doubleArr, typeInt,"clustered/part");
}else{
log.info("unclustered---->doubleArr:{},typeInt:{}",new Object[]{doubleArr,typeInt});
out.write("unclustered", doubleArr, typeInt,"unclustered/part");
}
} catch (Exception e) {
e.printStackTrace();
} finally {
IOUtils.closeStream(reader);
}
}
/**
* first select the smallest smallDistance if the types is not -1 then return
* else select the second smallest ,and so on
* at last ,return -1
* @param smallDistance
* @param types
* @return
*/
private int getTypeIndex(double[] smallDistance, int[] types) {
for (int i = 1; i < smallDistance.length; i++) {
for (int j = i; j > 0; j--) {
if (smallDistance[j] < smallDistance[j - 1]) {
swap(smallDistance,j,j-1);
swap(types,j,j-1);
} else
break;
}
}
for(int i=0;i<types.length;i++){
if(types!=-1){
return types;
}
}
return -1;
}
/**
* @param smallDistance
* @param j
* @param i
*/
private void swap(double[] smallDistance, int j, int i) {
double o = smallDistance[j];
smallDistance[j]=smallDistance;
smallDistance=o;
}
private void swap(int[] smallDistance, int j, int i) {
int o = smallDistance[j];
smallDistance[j]=smallDistance;
smallDistance=o;
}
/**
* @param smallDistance
* @param types
* @param distance
* @param type
*/
private void checkAndModify(double[] smallDistance, int[] types,
double distance, int type) {
double max= smallDistance[0];
int maxIndex =0;
for(int i=1;i<smallDistance.length;i++){
if(max<smallDistance){
maxIndex=i;
max=smallDistance;
}
}
if(max>distance){
smallDistance[maxIndex]=distance;
types[maxIndex]= type;
}
}
public void readCenterAndWrite(String centerPath){
}
@Override
public void cleanup(Context cxt){
try {
out.close();
} catch (IOException e) {
e.printStackTrace();
} catch (InterruptedException e) {
e.printStackTrace();
}
}
}[/mw_shl_code]
2. 操作步骤:拷贝target/fast_cluster-0.0.1-SNAPSHOT.jar包到集群$HADOOP_HOME/share/hadoop/mapreduce 目录下面; 拷贝src/main/resource/data/flume.csv 到集群目录 /user/root/flume.csv; 修改src/main/java/fz/utils/HUtils.java 的getConf方法,修改yarn.resourcemanager.address和yarn.resourcemanager.scheduler.address的地址; 打开src/test/java/fz/fast_cluster/ClusterDataDriverTest.java
1) 打开test_findCenter(input,dc,splitter,method); 的注释并注释掉test_clusterData(input,k,splitter);;
2)修改其中的input路径为实际路径;(如果数据有变,则需根据实际情况确定dc的值)
3)直接java运行,计算各个点的局部密度和最小大密度距离;
4)运行完成后可以在d:/decision_chart.png查看该决策图;
5)根据决策图人为确定聚类个数; 打开src/test/java/fz/fast_cluster/ClusterDataDriverTest.java
1) 注释掉test_findCenter(input,dc,splitter,method); 并打开注释掉的test_clusterData(input,k,splitter);;
2)修改其中的聚类个数K;
3)直接java运行,即可根据聚类中心进行分类; 修改src/test/java/fz/fast_cluster/HUtilsTest.java ;
1)修改路径localPath路径为本地存储各次循环确定聚类点的数据路径,修改iter_i(为聚类循环的次数);
2)运行完成后可以在localPath查看各次循环的分类好的数据; 未实现部分:
1)在寻找点局部密度时,给定的阈值dc人为确定,可以考虑使用程序确定;
2)聚类个数人为确定,同样可以考虑程序确定;
3)在根据聚类中心进行分类的时候,距离使用各个聚类中心点的最小距离的一半作为起始阈值,接着每次循环递增10%,直到递增到各个聚类中心点最大距离的1/3,这个可以优化;
4)暂不支持大数据或多文件;
3. 实验使用flume.csv的数据进行实验,得到的决策图如下所示:
循环一共运行了8次,每次的类别图如下所示:
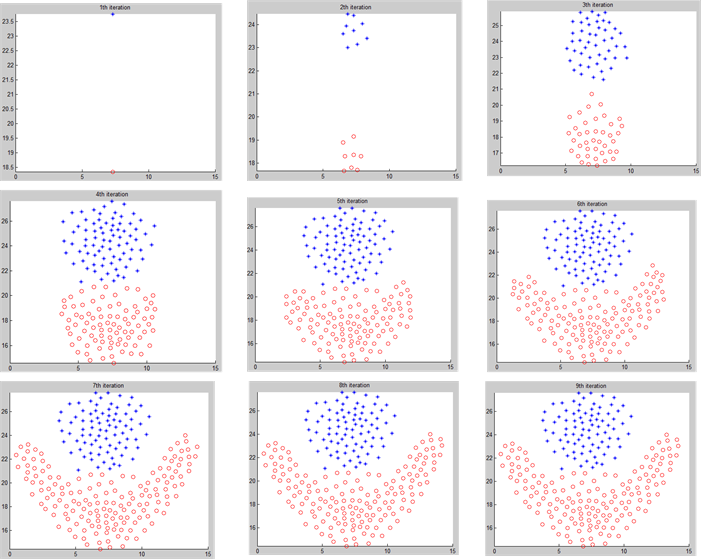
原始数据作图如下:
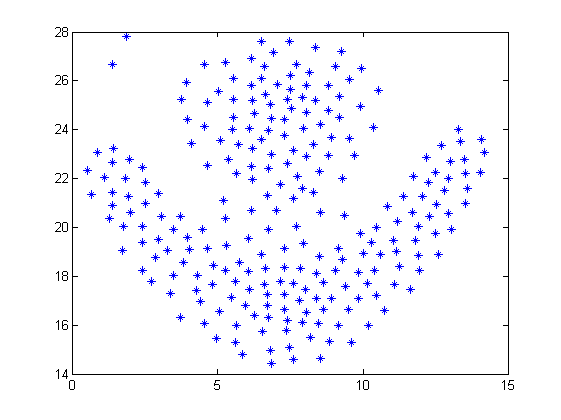
这里可以看到有两个点没有被分类,同时查看HDFS:
同样可以看到循环结束后,还有点没有被分类。这里其实也可以不用来分类,可以直接理解为异常点也可以。
分享,成长,快乐 脚踏实地,专注
|
 /2
/2 